Overexpression of lasB Gene in Klebsiella pneumoniae and its Effect on Biofilm Formation and Antibiotic Resistance
DOI:
https://doi.org/10.54133/ajms.v6i2.668الكلمات المفتاحية:
Antibiotic resistance، Biofilm formation، K. pneumoniaeالملخص
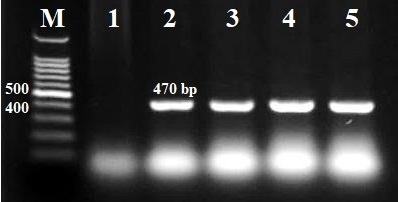
الخلفية: تعتبر الكلبسيلا الرئوية ثاني أكثر البكتيريا الممرضة شيوعًا في عائلة Enterobacteriaceae بعد الإشريكية القولونية. كما تعتبر واحدًا من الكائنات الدقيقة الممرضة المهمة التي تُسبب العدوى المرتبطة بالرعاية الصحية في جميع أنحاء العالم. الأهداف: دراسة تأثير الافراط في تعبير جين الإيلاستيز (lasB) في الكلبسيلا الرئوية على مقاومة المضادات الحيوية وتكوين الغشاء الحيوي. الطرق: تم الحصول على 25 عزلة سريرية من الكلبسيلا الرئوية من مركز التحليل الكيميائي (CAC) في بغداد، العراق وإعادة تشخيصها باستخدام نظام Vitek-2. تم استخدام طبق المايكروتيتر لقياس تكوين الغشاء الحيوي باستخدام اختبار ELISA. تم استخدام طريقة انتشار القرص في اختبارات الحساسية للمضادات الحيوية وفقًا لإرشادات CLSI 2022. تم ادخال البلازميد plasB إلى الكلبسيلا الرئوية باستخدام طريقة الادخال الكهربائي. النتائج: من بين 25 عزلة، كانت 11 (44%)، 11 (44%)، و3 (12%) منتجة للغشاء الحيوي بشكل قوي، متوسط، وضعيف على التوالي. تم اختيار إحدى منتجات الغشاء الحيوي القوية (KA1) للدراسة اللاحقة. تم ادخال بلازميد plasB بنجاح إلى KA1 لإنتاج عزلة KA1(plasB). كان تكوين الغشاء الحيوي لدى KA1(plasB) أقل بشكل كبير من KA1، كما أظهرت KA1(plasB) حساسية تجاه التتراسيكلين والدوكسيسيكلين وحمض الأموكسيسيلين-الكلافيولانيك بالمقارنة مع KA1. علاوة على ذلك، أظهرت KA1(plasB) انخفاضًا كبيرًا في مقاومتها للأمبيسيلين وزيادة كبيرة في حساسيتها تجاه سيبروفلوكساسين، بينما لم تظهر اختلافات في قابلية التحسس تجاه ليفوفلوكساسين، سيفوتاكسيم، بيبراسيلين-تازوباكتام، أميكاسين، وإيريثرومايسين بالمقارنة مع KA1. الاستنتاج: أظهرت هذه الدراسة أن الافراط في تعبير جين الإيلاستيز (lasB) له تأثيرات كبيرة على تكوين الغشاء الحيوي ومقاومة المضادات الحيوية في الكلبسيلا الرئوية.
التنزيلات
المراجع
Wang G, Zhao G, Chao X, Xie L, Wang H. The Characteristic of Virulence, Biofilm and Antibiotic Resistance of Klebsiella pneumoniae. Int J Environ Res Public Health. 2020;17(17):6278. doi: 10.3390/ijerph 17176278. DOI: https://doi.org/10.3390/ijerph17176278
Tabassum R, Shafique M, Khawaja KA, Alvi IA, Rehman Y, Sheik CS, et al. Complete genome analysis of a Siphoviridae phage TSK1 showing biofilm removal potential against Klebsiella pneumoniae. Sci Rep. 2018;17;8(1):17904. doi: 10.1038/s41598-018-36229-y. DOI: https://doi.org/10.1038/s41598-018-36229-y
Hussein MH, Aal Owaif HA, Abdulateef SA. The aminoglycoside resistance genes, pehX, blaCTX-M, blaAmpC, and npsB among Klebsiella oxytoca stool samples. Int J Biomed. 2023;13(3):127-130. doi: 10.21103/Article13(3)_OA13. DOI: https://doi.org/10.21103/Article13(3)_OA13
Nirwati H, Sinanjung K, Fahrunissa F, Wijaya F, Napitupulu S, Hati VP, et al. Biofilm formation and antibiotic resistance of Klebsiella pneumoniae isolated from clinical samples in a tertiary care hospital, Klaten, Indonesia. BMC Proc. 2019;13(Suppl 11):20. doi: 10.1186/s12919-019-0176-7. DOI: https://doi.org/10.1186/s12919-019-0176-7
Vuotto C, Longo F, Balice MP, Donelli G, Varaldo PE. Antibiotic Resistance Related to Biofilm Formation in Klebsiella pneumoniae. Pathogens. 2014;3(3):743-758. doi: 10.3390/pathogens3030743. DOI: https://doi.org/10.3390/pathogens3030743
Patel R. Biofilms and antimicrobial resistance. Clin Orthop Relat Res. 2005;(437):41-7. doi: 10.1097/01.blo.0000175714.68624.74. DOI: https://doi.org/10.1097/01.blo.0000175714.68624.74
Galdeno ACM, Branquinha MH, Santos AL, Viganor L, (Eds.), Pseudomonas aeruginosa and its arsenal of proteases: weapons to battle the host. In: Pathophysiological Aspects of Proteases; 2017. p. 381-397. doi: 10.1007/978-981-10-6141-7_16. DOI: https://doi.org/10.1007/978-981-10-6141-7_16
Cathcart GRA, Quinn D, Greer B, Harriott P, Lynas JF, Gilmore BF, et al. Novel inhibitors of the Pseudomonas aeruginosa virulence factor LasB: a potential therapeutic approach for the attenuation of virulence mechanisms in pseudomonal infection. Antimicrob Agents Chemother. 2011;55(6):2670-2678. doi: 10.1128/AAC.00776-10. DOI: https://doi.org/10.1128/AAC.00776-10
Jurado-Martín I, Sainz-Mejías M, McClean S. Pseudomonas aeruginosa: An audacious pathogen with an adaptable arsenal of virulence factors. Int J Mol Sci. 2021;18;22(6):3128. doi: 10.3390/ijms22063128. DOI: https://doi.org/10.3390/ijms22063128
Abidi SH, Sherwani SK, Siddiqui TR, Bashir A, Kazmi SU. Drug resistance profile and biofilm forming potential of Pseudomonas aeruginosa isolated from contact lenses in Karachi-Pakistan. BMC Ophthalmol. 2013;17;13:57. doi: 10.1186/1471-2415-13-57. DOI: https://doi.org/10.1186/1471-2415-13-57
Abdulateef SA, Al-Salmani MS, Aal Owaif HA. Acinetobacter baumannii producing ESBLs and carbapenemases in the Intensive Care Units developing fosfomycin and colistin resistance. J Appl Natural Sci. 2023;15(3):1263-1267. doi:10.31018/jans.v15i3.4872. DOI: https://doi.org/10.31018/jans.v15i3.4872
CLSI. Performance Standards for Antimicrobial Susceptibility Testing, M100 (32nd Ed.). Clinical and Laboratory Standards Institute; 2022.
Aal Owaif HA, Mhawesh AA, Abdulateef SA. The role of BipA in the regulation of K1 capsular polysaccharide production of uropathogenic Escherichia coli. Ann Trop Med Public Health. 2019;22 (Special Issue):S254. doi: 10.36295/ASRO.2019.220924. DOI: https://doi.org/10.36295/ASRO.2019.220924
Sambrook J, Russell DW, (Eds.), Molecular Cloning: A Laboratory Manual, (3rd Ed.), Vol. 1. Cold Spring Harbor Laboratory Press, New York; 2001.
Ullah A, Bashir A, Rehman B, Naeem W, Zara Shah S. Optimization of colony polymerase chain reaction for the 16S rRNA of different strains of Escherichia coli. Innovare J Life Sci. 2023;11(1):32-35. doi: 10.22159/ijls.2023.v11i1.48219. DOI: https://doi.org/10.22159/ijls.2023.v11i1.48219
Al-Dulaymi AAM, Aal Owaif HA. Antibiotic susceptibility and biofilm formation of Klebsiella pneumoniae. Dijlah J. 2023;6(4):326-335.
Al-Rubii BAL. Cloning LasB gene of Pseudomonas aeruginosa elastase 10104-2aI in E. coli BL21 and E. coli DH5α and investigated their effect on the stripping of Vero cells. Pak J Biotechnol. 2017;14(4):697-705.
Roshani-Asl P, Rashidi N, Shokoohizadeh L, Zarei J. Relationship among antibiotic resistance, biofilm formation and lasB gene in Pseudomonas aeruginosa isolated from burn patients. Clin Lab. 2018;64(9):1477-1484. doi: 10.7754/clin.lab.2018.180331. DOI: https://doi.org/10.7754/Clin.Lab.2018.180331
Mahdavi Z, Hemati S, Sadeghifard N, Jalilian FA, Taherikalani M, Bimanand L, et al. The Association between lasB and nanI genes with biofilm formation in Pseudomonas aeruginosa clinical isolates. J Clin Diagn Res. 2020;14(5):1-3. doi: 10.7860/JCDR/2020/16076.13697. DOI: https://doi.org/10.7860/JCDR/2020/16076.13697
Tielen P, Rosenau F, Wilhelm S, Jaeger KE, Flemming HC, Wingender J. Extracellular enzymes affect biofilm formation of mucoid Pseudomonas aeruginosa. Microbiology (Reading). 2010;156(Pt 7):2239-2252. doi: 10.1099/mic.0.037036-0. DOI: https://doi.org/10.1099/mic.0.037036-0
Abd Al-Rhman RM, Al-Aubydi MA. Determination the relationship between some genetic aspects with the capsule formation for pathogenic Klebsiella pneumoniae serotypes K1 & K2. Iraqi J Sci. 2015;56(2B):1385-1393.
Jabar AAA, Abid IN. Detection of CTX-M and Carbapenem hydrolyzing Beta-lactamase KPC in clinical isolates of Klebsiella pneumoniae. Ann R.S.C.B. 2021;25(4):876-887.
Al-Sheboul SA, Al-Madi GS, Brown B, Hayajneh WA. Prevalence of extended-spectrum β-lactamases in multidrug-resistant Klebsiella pneumoniae isolates in Jordanian hospitals. J Epidemiol Glob Health. 2023;13(2):180-190. doi: 10.1007/s44197-023-00096-2. DOI: https://doi.org/10.1007/s44197-023-00096-2
Abbas A. Antibacterial activities of various antibiotics against Klebsiella pneumoniae in clinical isolates. Pak BioMed J. 2023;6(01):18-21. doi: 10.54393/pbmj.v6i01.844. DOI: https://doi.org/10.54393/pbmj.v6i01.844
Raouf FE, Benyagoub E, Alkhudhairy MK, Akrami S, Saki M. Extended-spectrum beta-lactamases among Klebsiella pneumoniae from Iraqi patients with community-acquired pneumonia. Rev Assoc Med Bras. 2022;24;68(6):833-837. doi: 10.1590/1806-9282.20220222. DOI: https://doi.org/10.1590/1806-9282.20220222
Li Y, Kumar S, Zhang L, Wu H, Wu H. Characteristics of antibiotic resistance mechanisms and genes of Klebsiella pneumoniae. Open Med (Wars). 2023;18(1):20230707. doi: 10.1515/med-2023-0707. DOI: https://doi.org/10.1515/med-2023-0707
Bokaeian M, Saeidi S, Shahi Z, Kadaei V. tetA and tetB genes in Klebsiella pneumoniae isolated from clinical samples. Gene Cell Tissue. 2014;1(2):e18152. doi: 10.17795/gct-18152. DOI: https://doi.org/10.17795/gct-18152
Safika S, Nilasari Z, Pasaribu FHP. Detection of antibiotic resistance coding gene in Klebsiella pneumoniae bacteria isolated from broiler chickens in West Java, Indonesia. J Appl Pharm Sci. 2022;12(07):190-198. doi: 10.7324/JAPS.2022.120719. DOI: https://doi.org/10.7324/JAPS.2022.120719
Alansary IMM, Al-Saryi NA. Detection of biofilm formation in classical and hypervirulent Klebsiella pneumoniae. Al-Mustansiriyah J Sci. 2023;33(5):65-71. doi: 10.23851/mjs.v33i5.1315. DOI: https://doi.org/10.23851/mjs.v33i5.1315
Sweedan EG, Shehab ZH, Flayyih MT. Effect of gentamicin and doxycycline on expression of relB and relE genes in Klebsiella pneumonia. J Adv Biotechnol Exp Ther. 2022;5(3):667-675. doi: 10.5455/jabet.2022.d145. DOI: https://doi.org/10.5455/jabet.2022.d145
Ghanem S, El Shafey HM, Tamer EKA, Manzoor N. Antimicrobial resistance patterns of Klebsiella isolates from clinical samples in a Saudi hospital. Afr J Microbiol Res. 2017;11(23):965-971. doi: 10.5897/AJMR2017.8451. DOI: https://doi.org/10.5897/AJMR2017.8451
Akter T, Khan S, Nahar S, Fatema K, Shaha M, Hossain MJ, et al. Determination of prevalence and antimicrobial sensitivity patterns of Klebsiella pneumoniae from sputum sample of a tertiary care hospital. Int Med J. 2019;24(3):29-35.
Ahmadi M, Ranjbar R, Behzadi P, Mohammadian T. Virulence factors, antibiotic resistance patterns, and molecular types of clinical isolates of Klebsiella pneumoniae. Expert Rev Anti Infect Ther. 2022;20(3):463-472. doi: 10.1080/14787210.2022.1990040. DOI: https://doi.org/10.1080/14787210.2022.1990040
Owaif HAA, Aldulaimy MK, Abdulateef SA. The antibiotic resistance genes blaSHV, blaOXA-48, blaTEM and blaIMP in Pseudomonas aeruginosa isolated from respiratory tract infections in Baghdad, Iraq. Int J Biomed. 2023;13(4):341-344. doi: 10.21103/Article13(4)_OA18. DOI: https://doi.org/10.21103/Article13(4)_OA18
التنزيلات
منشور
كيفية الاقتباس
إصدار
القسم
الرخصة
الحقوق الفكرية (c) 2024 Al-Rafidain Journal of Medical Sciences

هذا العمل مرخص بموجب Creative Commons Attribution-NonCommercial-ShareAlike 4.0 International License.
Published by Al-Rafidain University College. This is an open access journal issued under the CC BY-NC-SA 4.0 license (https://creativecommons.org/licenses/by-nc-sa/4.0/).










